Python: Dizionari¶
I dizionari rappresentano mappe tra oggetti: mappano una chiave nel valore corrispondente.
Avvertimento
I dizionari sono mutabili!
Per definire un dizionario scrivo:
codice = {
"UUU": "F", # fenilalanina
"UCU": "S", # serina
"UAU": "Y", # tirosina
"UGU": "C", # cisteina
"UUC": "F", # fenilalanina
"UCC": "S", # serina
"UAC": "Y", # tirosina
# ...
}
Qui codice implementa il codice genetico, che mappa da stringhe di tre
lettere (le chiavi) al nome dell’aminoacido corrispondente (i valori).
La sintassi e’:
{ chiave1: valore1, chiave2: valore2, ...}
Uso un dizionario in questo modo:
aa_di_uuu = codice["UUU"]
print aa_di_uuu # "phenylalanine"
aa_di_ucu = codice["UCU"]
print aa_di_ucu # "serine"
Posso usare questo dizionario per “simulare” il processo della traduzione. Ad esempio, partendo da una sequenza di mRNA:
rna = "UUUUCUUAUUGUUUCUCC"
la spezzo in triplette:
triplette = [rna[i:i+3] for i in range(0, len(rna), 3)]
ottenendo:
>>> print triplette
['UUU', 'UCU', 'UAU', 'UGU', 'UUC', 'UCC']
ed a questo punto posso fare:
proteina = "".join([codice[tripletta] for tripletta in triplette])
ottenendo:
>>> print proteina
"FSYCFS"
La differenza piu’ notevole rispetto alla vera traduzione e’ che il nostro codice non rispetta i codoni di terminazione, ma e’ un difetto correggibile.
Avvertimento
Le chiavi non possono essere ripetute, i valori si’!
Nell’esempio sopra, ogni chiave e’ una tripletta ed e’ associata ad un unico valore, ma lo stesso valore puo’ essere associato a piu’ chiavi:
print codice["UCU"] # "S", serina
print codice["UCC"] # "S", serina
Esempio. Creo un dizionario che mappa ogni aminoacido nel suo volume (approssimato, in Amstrong cubici):
volume_di = {
"A": 67.0, "C": 86.0, "D": 91.0,
"E": 109.0, "F": 135.0, "G": 48.0,
"H": 118.0, "I": 124.0, "K": 135.0,
"L": 124.0, "M": 124.0, "N": 96.0,
"P": 90.0, "Q": 114.0, "R": 148.0,
"S": 73.0, "T": 93.0, "V": 105.0,
"W": 163.0, "Y": 141.0,
}
# Stampo il volume della citosina
print volume_di["C"] # 86.0
print type(volume_di["C"]) # float
Qui le chiavi sono di tipo str (immutabili) ed i valori sono dei float
(sempre immutabili).
Avvertimento
Non ci sono restrizioni sul tipo degli oggetti che si possono usare come valori. Come chiavi invece si possono usare solo oggetti immutabili!
Riassumo in che modo possono essere usati i vari tipi:
| Tipo | Come chiavi | Come valori |
|---|---|---|
| bool | ✓ | ✓ |
| int | ✓ | ✓ |
| float | ✓ | ✓ |
| str | ✓ | ✓ |
| list | NO | ✓ |
| tuple | ✓ | ✓ |
| dict | NO | ✓ |
Si veda l’esempio seguente.
Esempio. Creo un dizionario che mappa da ogni aminoacido ad una lista di due proprieta’: massa e volume:
proprieta_di = {
"A": [ 89.09, 67.0],
"C": [121.15, 86.0],
"D": [133.10, 91.0],
"E": [147.13, 109.0],
# ...
}
# Stampo massa e volume dell'alanina
print proprieta_di["A"] # [89.09, 67.0]
print type(proprieta_di["A"]) # list
Qui le chiavi sono str (immutabili) ed i valori sono list (mutabili).
Provo a creare il dizionario inverso (limitandomi al primo elemento per semplicita’):
da_proprieta_ad_aa = { [89.09, 67.0]: "A" }
Mi aspetto che questo dizionario mappi dalla lista:
[89.09, 67.0]
ad "A". Pero’ quando provo a crearlo Python da’ errore:
Traceback (most recent call last):
File "<stdin>", line 1, in <module>
TypeError: unhashable type: 'list'
# ^^^^^^^^^^^^^^^^^^^^^^^
# list e' mutabile!
Questo succede perche’ le chiavi hanno tipo list, che non e’ immutabile!
Per risolvere il problema posso usare, al posto di liste, delle tuple:
da_proprieta_ad_aa = {
( 89.09, 67.0): "A",
(121.15, 86.0): "C",
(133.10, 91.0): "D",
(147.13, 109.0): "E",
# ...
}
aa = da_proprieta_ad_aa[(133.10, 91.0)]
print aa # "D"
print type(aa) # str
Ora le chiavi sono tuple, immutabili, e va tutto bene.
Operazioni¶
| Ritorna | Operatore | Significato |
|---|---|---|
int |
len(dict) |
Restituisce il numero di coppie chiave-valore |
object |
dict[object] |
Restituisce il valore associato ad una chiave |
| – | dict[object]=object |
Inserisce o sostituisce una coppia chiave-valore |
Esempio. Partendo dal dizionario vuoto:
codice = {}
print codice # {}
print len(codice) # 0
ricreo il dizionario del primo esempio inserendo a mano tutte le coppie chiave-valore:
codice["UUU"] = "F" # fenilalanina
codice["UCU"] = "M" # metionina
codice["UAU"] = "Y" # tirosina
# ...
print codice
print len(codice)
Mi accorgo di avere fatto un errore qui sopra: "UCU" dovrebbe corrispondere
a "S" (serina), non a "M" (metionina). Come faccio a correggere l’errore?
Risposta: sostituendo il valore corrispondente alla chiave "UCU" con quello
corretto:
codice["UCU"] = "S" # serina
Qui alla chiave "UCU", che gia’ era nel dizionario, associo un nuovo valore
"S" (serina).
Avvertimento
Se provo ad ottenere il valore di una chiave non presente nel dizionario:
>>> codice[":-("]
Python da’ errore:
Traceback (most recent call last):
File "<stdin>", line 1, in <module>
KeyError: ":-("
# ^^^^^
# questa e' la chiave che non esiste
Metodi¶
| Ritorna | Metodo | Significato |
|---|---|---|
object |
dict.get(k, [default]) |
Resituisce il valore della chiave, oppure il default |
bool |
dict.has_key(object) |
True se la chiave e’ nel dizionario |
list |
dict.keys() |
Restituisce una lista delle chiavi |
list |
dict.values() |
Restituisce una lista dei valori |
list-of-tuples |
dict.items() |
Restituisce una lista di coppie chiave-valore |
Esempio. Partendo dal dizionario codice definito nel primo esercizio:
codice = {
"UUU": "F", # fenilalanina
"UCU": "S", # serina
"UAU": "Y", # tirosina
"UGU": "C", # cisteina
"UUC": "F", # fenilalanina
"UCC": "S", # serina
"UAC": "Y", # tirosina
# ...
}
posso ottenere le chiavi cosi’:
>>> chiavi = codice.keys()
>>> print chiavi
["UUU", "UCU", "UAU", ...]
ed i valori cosi’:
>>> valori = codice.values()
>>> print valori
["F", "S", Y", "C", ...]
oppure sia chiavi che valori cosi’:
>>> coppie_chiave_valore = codice.items()
>>> print coppie_chiave_valore
[("UUU", "F"), ("UCU", "S"), ...]
Infine posso controllare se una certa chiave sta nel dizionario:
>>> print codice.has_key("UUU")
True
>>> print codice.has_key(":-(")
False
Avvertimento
Non c’e’ alcuna garanzia che un dizionario preservi l’ordine in cui vengono inseriti gli elementi.
Ad esempio:
>>> d = {}
>>> d["z"] = "zeta"
>>> d["a"] = "a"
>>> d
{'a': 'a', 'z': 'zeta'}
>>> d.keys()
['a', 'z']
>>> d.values()
['a', 'zeta']
>>> d.items()
[('a', 'a'), ('z', 'zeta')]
Qui ho inserito prima "z" e poi "a", ma quando estraggo dal
dizionario d le chiavi, i valori e le coppie chiave-valore,
l’ordine in cui mi restituisce "z" ed "a" e’ invertito!
Esempio. Posso usare un dizionario per rappresentare un oggetto strutturato, ad esempio le proprieta’ di una catena proteica:
chain = {
"name": "1A3A",
"chain": "B",
"sequence": "MANLFKLGAENIFLGRKAATK...",
"num_scop_domains": 4,
"num_pfam_domains": 1,
}
print chain["name"]
print chain["sequence"]
print chain["num_scop_domains"]
Scrivere a mano dizionari come questo e’ sconveniente, ma quando leggeremo informazioni da file (possibilmente da interi database biologici), avere sotto mano dizionari fatti cosi’ puo’ essere molto comodo.
(Soprattutto se sopra ci costruiamo algoritmi per analizzare in modo automatico i dati!)
Esempio. Dato il sequente testo in FASTA (accorciato per motivi di spazio) che descrive la sequenza primaria della retrotranscriptasi del virus HIV-1:
>2HMI:A|PDBID|CHAIN|SEQUENCE
PISPIETVPVKLKPGMDGPKVKQWPLTEEKIKALVEICTEMEKEGKISKI
NKRTQDFWEVQLGIPHPAGLKKKKSVTVLDVGDAYFSVPLDEDFRKYTAF
QSSMTKILEPFKKQNPDIVIYQYMDDLYVGSDLEIGQHRTKIEELRQHLL
VQPIVLPEKDSWTVNDIQKLVGKLNWASQIYPGIKVRQLSKLLRGTKALT
PSKDLIAEIQKQGQGQWTYQIYQEPFKNLKTGKYARMRGAHTNDVKQLTE
WWTEYWQATWIPEWEFVNTPPLVKLWYQLEKEPIVGAETFYVDGAANRET
AIYLALQDSGLEVNIVTDSQYALGIIQAQPDKSESELVNQIIEQLIKKEK
>2HMI:B|PDBID|CHAIN|SEQUENCE
PISPIETVPVKLKPGMDGPKVKQWPLTEEKIKALVEICTEMEKEGKISKI
NKRTQDFWEVQLGIPHPAGLKKKKSVTVLDVGDAYFSVPLDEDFRKYTAF
QSSMTKILEPFKKQNPDIVIYQYMDDLYVGSDLEIGQHRTKIEELRQHLL
VQPIVLPEKDSWTVNDIQKLVGKLNWASQIYPGIKVRQLSKLLRGTKALT
PSKDLIAEIQKQGQGQWTYQIYQEPFKNLKTGKYARMRGAHTNDVKQLTE
WWTEYWQATWIPEWEFVNTPPLVKLWYQLE
>2HMI:C|PDBID|CHAIN|SEQUENCE
DIQMTQTTSSLSASLGDRVTISCSASQDISSYLNWYQQKPEGTVKLLIYY
EDFATYYCQQYSKFPWTFGGGTKLEIKRADAAPTVSIFPPSSEQLTSGGA
NSWTDQDSKDSTYSMSSTLTLTADEYEAANSYTCAATHKTSTSPIVKSFN
>2HMI:D|PDBID|CHAIN|SEQUENCE
QITLKESGPGIVQPSQPFRLTCTFSGFSLSTSGIGVTWIRQPSGKGLEWL
FLNMMTVETADTAIYYCAQSAITSVTDSAMDHWGQGTSVTVSSAATTPPS
TVTWNSGSLSSGVHTFPAVLQSDLYTLSSSVTVPSSTWPSETVTCNVAHP
>2HMI:E|PDBID|CHAIN|SEQUENCE
ATGGCGCCCGAACAGGGAC
>2HMI:F|PDBID|CHAIN|SEQUENCE
GTCCCTGTTCGGGCGCCA
Le sequenza e’ presa dalla Protein Data Bank, struttura 2HMI.
Posso immaginare di mettere questa informazione in un dizionario:
sequenze_2HMI = {
"A": "PISPIETVPVKLKPGMDGPKVKQWPLTEEKI...",
"B": "PISPIETVPVKLKPGMDGPKVKQWPLTEEKI...",
"C": "DIQMTQTTSSLSASLGDRVTISCSASQDISS...",
"D": "QITLKESGPGIVQPSQPFRLTCTFSGFSLST...",
"E": "ATGGCGCCCGAACAGGGAC",
"F": "GTCCCTGTTCGGGCGCCA",
}
Dato questo dizionario posso estrarre facilmente la sequenza di ogni singola catena della struttura 2HMI:
>>> print sequenze_2HMI["F"]
"GTCCCTGTTCGGGCGCCA"
calcolare di quante catene e’ composta la struttura:
num_catene = len(sequenze_2HMI)
calcolare statistiche sulla co-occorrenza di aminoacidi, provare a predire le strutture secondarie, allineare le sequenze contro qualche database...
Esempio. I dizionari sono utili anche per descrivere istogrammi. Ad esempio, supponiamo di avere una sequenza:
seq = "GTCCCTGTTCGGGCGCCA"
Calcolo le proporzioni dei vari nucleotidi:
num_A = seq.count("A") # 1
num_T = seq.count("T") # 4
num_C = seq.count("C") # 7
num_G = seq.count("G") # 6
Voglio catturare questa informazione statistica in un istogramma. Lo implemento cosi’:
istogramma = {
"A": float(num_A) / len(seq), # 1 / 18 ~ 0.06
"T": float(num_T) / len(seq), # 4 / 18 ~ 0.22
"C": float(num_C) / len(seq), # 7 / 18 ~ 0.38
"G": float(num_G) / len(seq), # 6 / 18 ~ 0.33
}
A questo punto posso recuperare la proporzione dei vari nucelotidi dall’istogramma:
prop_A = istogramma["A"]
print prop
Posso anche verificare che l’istogramma definisca una distribuzione di probabilita’ (approssimata), cioe’ che la somma delle proporzioni dia 1:
print istogramma["A"] + istogramma["C"] + ...
Esempio. Possiamo codificare una rete (in questo esempio fittizia) di interazioni fisiche o funzionali tra proteine cosi’:
partners_di = {
"2JWD": ("1A3A",),
"1A3A": ("2JWD", "2ZTI"),
"2ZTI": ("1A3A", "3BLF"),
"3BLU": ("1A3A", "3BLF"),
"3BLF": ("3BLU", "2ZTI"),
}
che rappresenta la rete:
2JWD ---------- 1A3A ---------- 2ZTI
| |
| |
3BLU ---------- 3BLF
Qui partners_di["1A3A"], ad esempio, e’ una tupla dove abbiamo messo tutti
i binding partners della proteina 1A3A.
Possiamo usare questo dizionario per trovare tutti i binding partners dei binding partners di 1A3A, e oltre:
# Estraggo i partner di 1A3A
partners = partners_di["1A3A"]
# Estraggo i partner dei partner di 1A3A
partners_2_step = partners_di[partners[0]] + \
partners_di[partners[1]] + \
...
partners_di[partners[-1]]
# Estraggo i parner dei partner dei partner di 1A3A
partners_3_step = partners_di[partners_2_step[0]] + \
partners_di[partners_2_step[1]] + \
...
partners_di[partners_2_step[-1]]
La stessa struttura si puo’ usare per codificare reti sociali (Facebook, Twitter, Google+, etc.) e trovare chi e’ amico (o segue) chi, o per individuare comunita’ di persone che si conoscono tra loro.
Nota
Ci sono molti altri modi di codificare reti. Un’alternativa al dizionario di cui sopra, e’ la matrice di adiacenza, che si puo’ implementare come una lista di liste.
Esercizi¶
Creare a mano:
Un dizionario vuoto. Controllare che sia vuoto con
len().Un dizionario
pronomiche rappresenta queste corrispondenze:1 -> "io" 2 -> "tu" 3 -> "egli" ...
Crearlo in due modi:
- In una sola riga di codice.
- Partendo da un dizionario vuoto ed aggiungendo passo passo tutte le coppie chiave valore.
Un dizionario
decuplo_diche implementa la funzione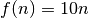 , che
associa ogni chiave (un intero) al suo decuplo.
, che
associa ogni chiave (un intero) al suo decuplo.Le chiavi devono essere gli interi da 1 a 5.
Una volta costruito il dizionario, applicarlo a tutti gli elementi di
range(2, 5)con una list comprehension e stampare a schermo il risultato.Poi fare la stessa cosa, pero’ per tutte le chiavi di
decuplo_di.Hint: la lista delle chiavi si puo’ ottenere con
keys().Un dizionario
info_2TMVche codifica informazioni strutturate sulla struttura PDB 2TMV della capside del Tobacco Mosaic Virus.- Alla chiave
"pdb_id"associate il valore"2TMV". - Alla chiave
"uniprot_id"associate il valore"P69687 (CAPSD_TMV)". - Alla chiave
"numero_domini_scp"associate1. - Alla chiave
"numero_domini_pfam"associate1.
- Alla chiave
Un dizionario
parenti_diche rappresenta questa rete:GIULIA ---- FRANCO ---- MATTEO | | + ----- BENEDETTA ------+
come nell’esempio visto in precedenza.
Una volta costruito il dizionario, stampare a schermo il numero di parenti di
"GIULIA".Un dizionario
da_2_bit_a_interoche rappresenta la mappa dalle seguenti coppie di interi ad intero:0, 0 -> 0 # 0*2**1 + 0*2**0 = 0 0, 1 -> 1 # 0*2**1 + 1*2**0 = 1 1, 0 -> 2 # 1*2**1 + 0*2**0 = 2 1, 1 -> 3 # 1*2**1 + 1*2**1 = 3 ^^^^ ^ ^^^^^^^^^^^^^^^^^^^ chiave valore spiegazione
Una volta creato il dizionario, stampare a schermo il valore corrispondente ad una delle quattro chiavi date (a scelta).
Dato:
rapporti = { ("A", "T"): 10.0 / 5.0, ("A", "C"): 10.0 / 7.0, ("A", "G"): 10.0 / 6.0, ("T", "C"): 5.0 / 7.0, ("T", "G"): 5.0 / 6.0, ("C", "G"): 7.0 / 6.0, }
che rappresenta rapporti tra il numero di A, T, C, e G in una sequenza:
Che differenza c’e’ tra
len(rapporti),len(rapporti.keys()),len(rapporti.values())elen(rapporti.items())?Controllare se
rapporticontiene la chiave("T", "A"). E la chiave("C", "G")?Hint: posso usare
keys()? Posso usare un altro metodo?Controllare se contiene il valore 2. E il valore 3?
Hint: posso usare
values()?Controllare se contiene la coppia chiave-valore
(("A", "T"), 2). E la coppia chiave-valore(("C", "G"), 1000)?Hint: posso usare
items()?Usare una list comprehension per estrarre le chiavi dal risultato di
items(). Poi fare la stessa cosa con le chiavi.
Dato:
mappa = { "zero": 1, "uno": 2, "due": 4, "tre": 8, "quattro": 16, "cinque": 32, }
Concatenare tutte le chiavi di
mappa, separate da spazi, in una sola stringastringa_delle_chiavi.Concatenare tutti i valori di
mappacome stringhe, separate da spazi, in una sola stringastringa_dei_valori.Hint: occhio che i valori di
mappanon sono stringhe!Mettere in una lista tutte le chiavi di
mappa.Mettere in una lista tutte le chiavi di
mappa, ordinate alfanumericamente.Hint: la lista restituita da
keys()e’ ordinata?Mettere in una lista tutti i valori di
mappa, ordinati in base all’ordine delle chiavi corrispondenti.Hint: come faccio ad ordinare una lista in base all’ordine di un’altra lista?
Dato:
traduzione_di = {"a": "ade", "c": "cyt", "g": "gua", "t": "tym"}
tradurre la lista:
lista = ["A", "T", "T", "A", "G", "T", "C"]
nella stringa:
"ade tym tym ade gua tym cyt"Hint: occhio che le chiavi del dizionario sono minuscole, mentre gli elementi di
listasono maiuscoli! Partite assumendo che non lo siano, poi modificate il codice per tenere conto di questa idiosincrasia.